Physique de la matière active et du génome
Nous explorons les principes physiques qui organisent la matière vivante — de l’intérieur des cellules à l’architecture tridimensionnelle du génome — pour comprendre comment émergent les fonctions biologiques.
L’équipe étudie la physique de la matière active et biologique et la physique du génome. Nous cherchons à comprendre comment des lois de la physique (transitions et séparations de phase, dynamiques hors équilibre, interactions collectives) façonnent l’organisation spatiale et temporelle des systèmes vivants.
Nos travaux portent sur :
- l’organisation intracellulaire à grande échelle ;
- l’architecture 3D du génome eucaryote et son lien avec l’épigénétique (répression, activation, expression des gènes) ;
- la ségrégation et le positionnement du génome bactérien via des mécanismes actifs (hors équilibre thermodynamique);
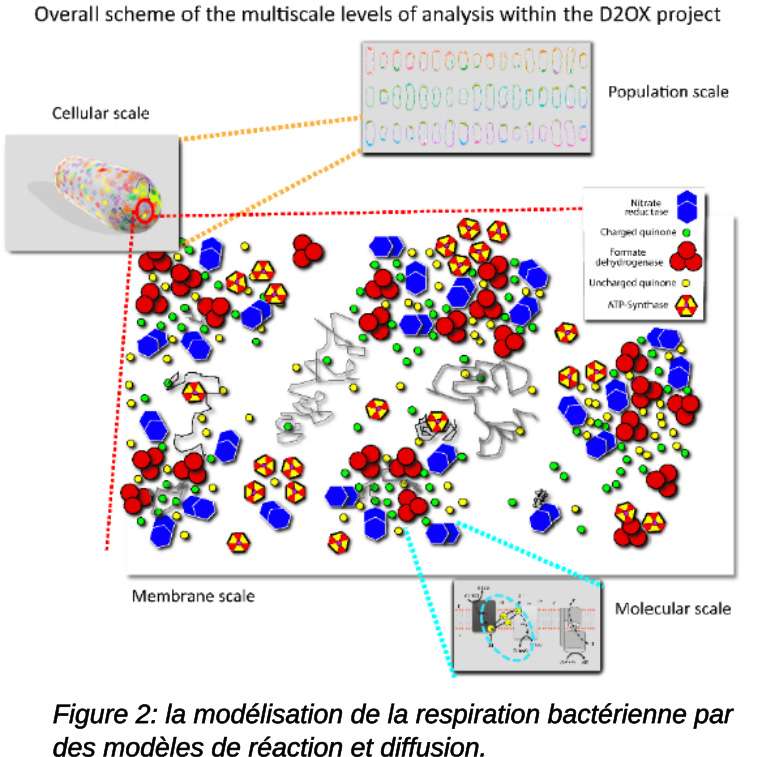
- la dynamique et la distribution de complexes bioénergétiques dans la membrane bactérienne;
- la modélisation de systèmes immunitaires vus comme des systèmes complexes et décrits en terme de dynamique des populations des cellules.
Nous combinons modélisation théorique, simulations numériques et analyse de données issues de techniques expérimentales de pointe (par ex. cartographie de contacts chromosomiques et imagerie multipoints) pour relier structure, dynamiques et fonction des systèmes étudiés.
Sous-thèmes :
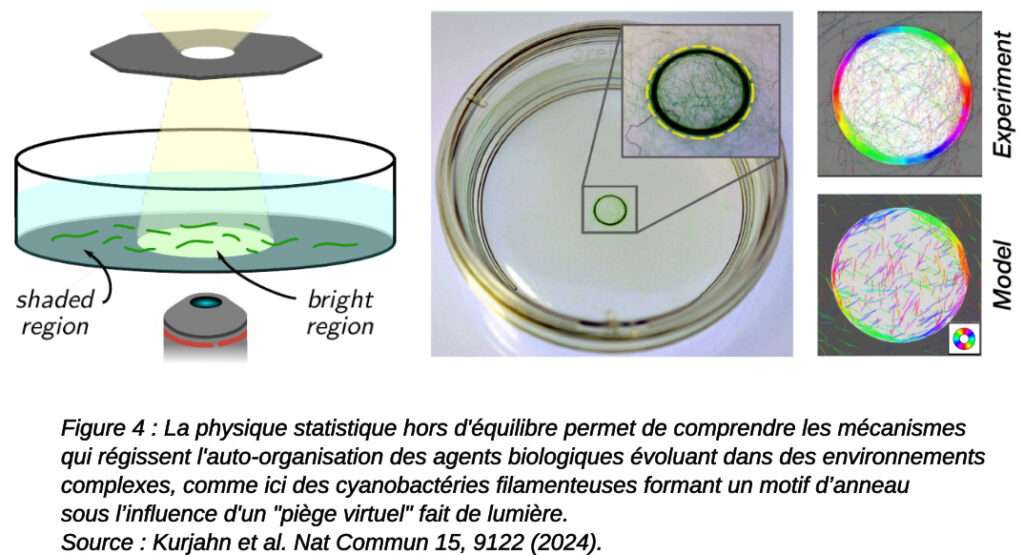
- Matière active & organisation intracellulaire : lois de l’auto-organisation et dynamiques hors équilibre de la matière biologique.
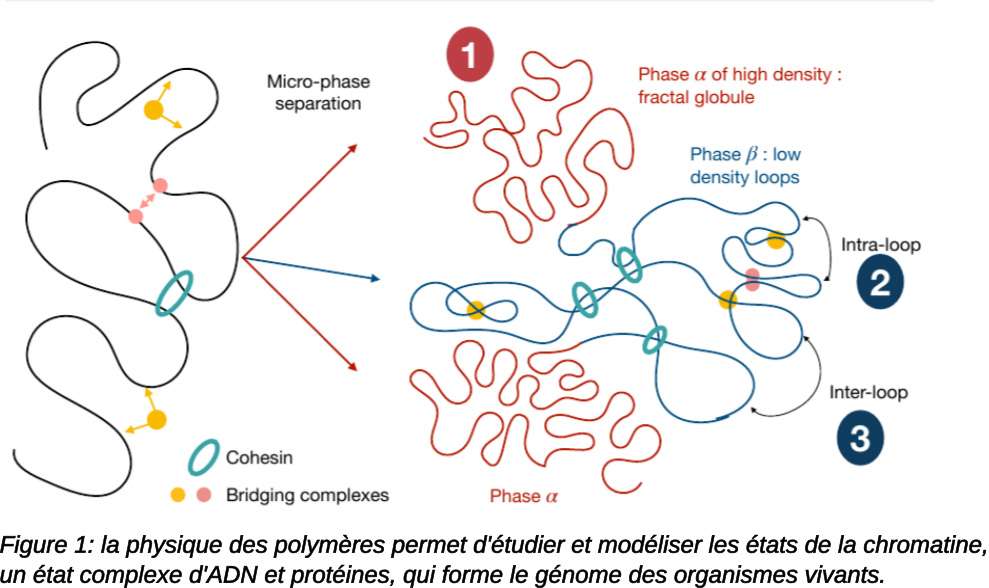
- Physique du génome : modèles issus de la physique statistique et des polymères reliant l’architecture tridimensionnelle du génome des cellules eucaryotes, l’épigénétique et les mécanismes d’expression et régulation des gènes.
- Génome bactérien : mécanismes actifs de ségrégation et de positionnement du génome bactérien permettant la division cellulaire.
- Nanobiophysique des membranes : distribution et dynamique de complexes protéiques impliqués dans l’établissement de la force protomotrice transmembranaire et la bioénergétique bactérienne.
- Systèmes immunologiques : modèles de populations et sur réseaux pour éclairer l’évolution et la dynamique de maladies (application en maladies auto-immunes comme l’arthrose rhumatoïde).
Méthodes :
Modélisation (équations stochastiques, méthodes variationnelles, systèmes d’équations différentielles), simulations (Monte Carlo, dynamique moléculaire/brownienne), analyse quantitative de données expérimentales