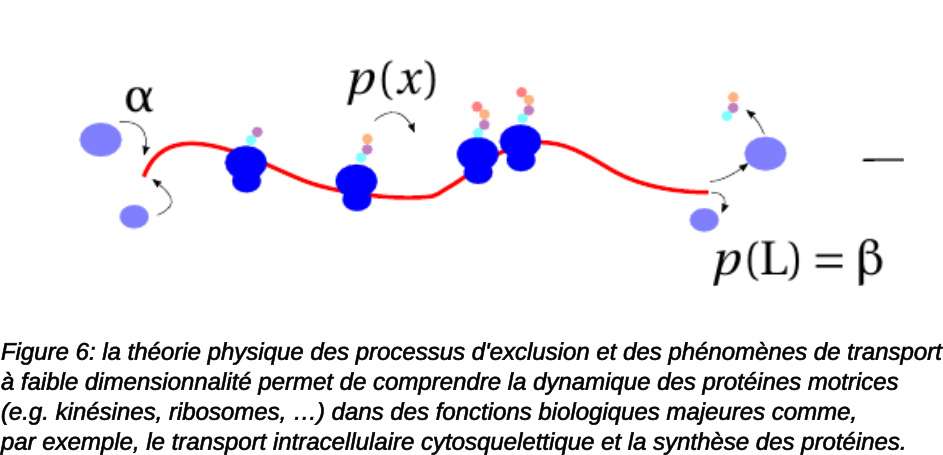
Transport unidirectionnel et traduction des gènes
Nous utilisons des modèles physiques pour comprendre comment les ribosomes traduisent l’ARN messager (ARNm) en protéines, un processus clé de la vie.
La traduction de l’ARNm par les ribosomes est une étape essentielle de l’expression des gènes. Pour la modéliser, nous utilisons des modèles issus de la physique statistique, en particulier le TASEP (Totally Asymmetric Simple Exclusion Process), un paradigme pour décrire le transport unidirectionnel de particules avec des interaction d’exclusion.
Ces modèles permettent d’explorer comment les ribosomes se déplacent le long de l’ARNm, comment les vitesses de traduction varient selon les codons, et comment des phénomènes comme la dégradation de l’ARNm influencent la production de protéines.
Nous développons des approches théoriques et numériques pour relier les profils de densité des ribosomes (issus de données Ribo-Seq) aux taux cinétiques sous-jacents, ouvrant la voie à une compréhension quantitative des mécanismes de régulation de la traduction ou comment le processus de transport intracellulaire des complexes ribosomaux couplés à la synthèse des protéines déterminent la dynamique spatio-temporelle des profils de concentration intracellulaire des protéines .
Sous-thèmes :
- Modèles stochastiques pour la traduction de l’ARN messager
- Impact des fluctuations et de la dégradation de l’ARN messager
- Méthodes inverses pour estimer les taux cinétiques
- Modèles de réaction et transport pour étudier l’organisation spatio-temporelle des protéines.